1. 用途
从Uniprot和RCSB PDB数据库中搜索蛋白记录,获取PDB结构。
2. 入口
平台地址:https://cloud.yinfotek.com/
功能入口:左侧菜单栏【计算方案】->【小工具】->【分子结构】->【搜索蛋白质】
3. 步骤
-
搜索Uniprot蛋白记录
有两种搜索方式:
-
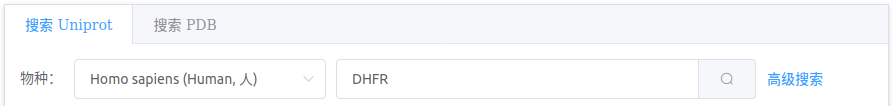
简单搜索
选择
物种,输入任意关键词,点击查询图标
-
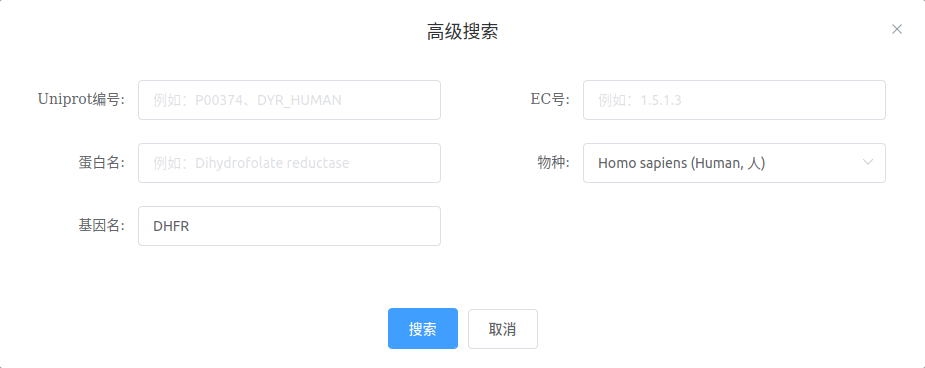
高级搜索
选择
高级搜索,填写选项,点击【搜索】。
-
-
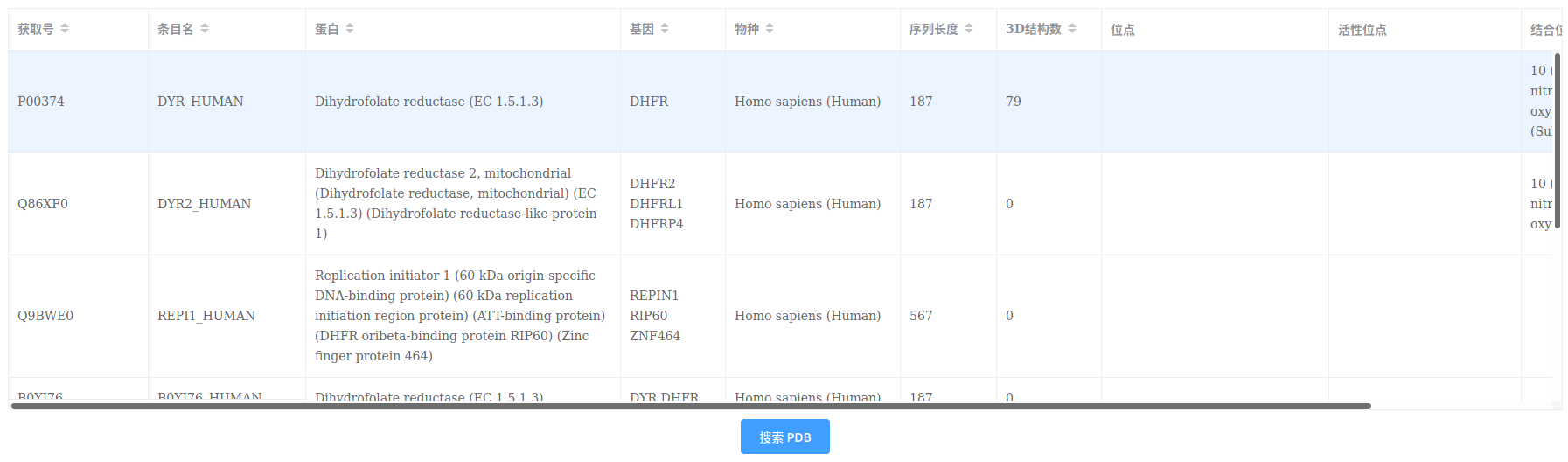
搜索PDB蛋白结构
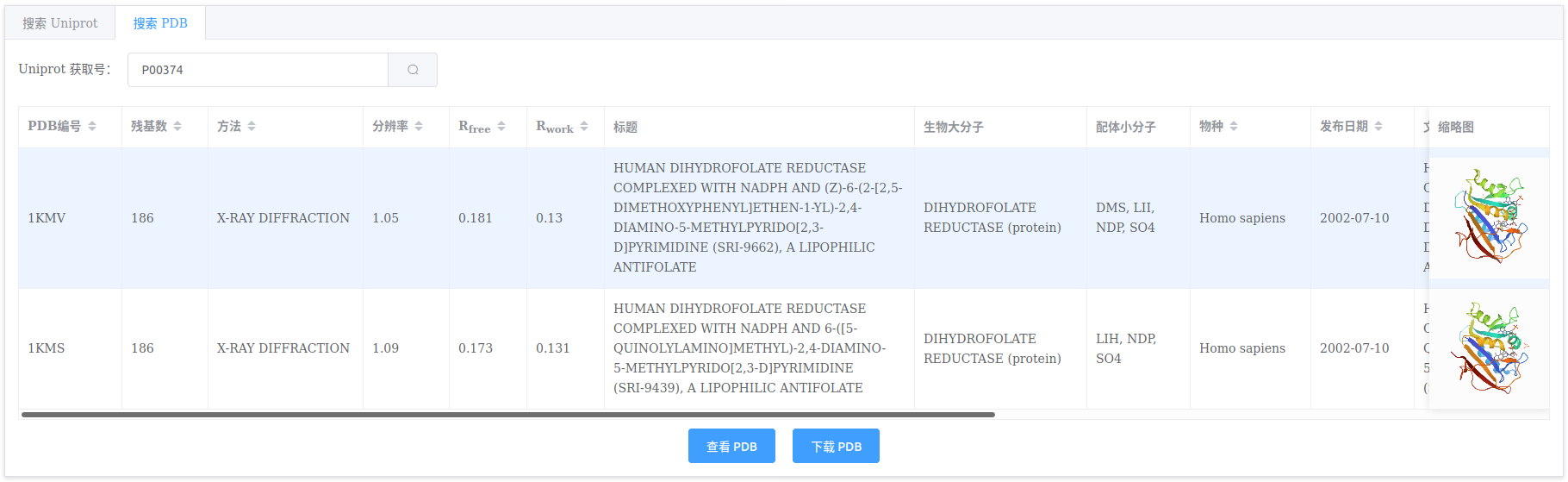
从Uniprot记录表中选择符合研究实际的蛋白记录,点击【搜索PDB】。
若已知Uniprot获取号(AC),可直接切换到【搜索PDB】页面,输入编号进行搜索。
-
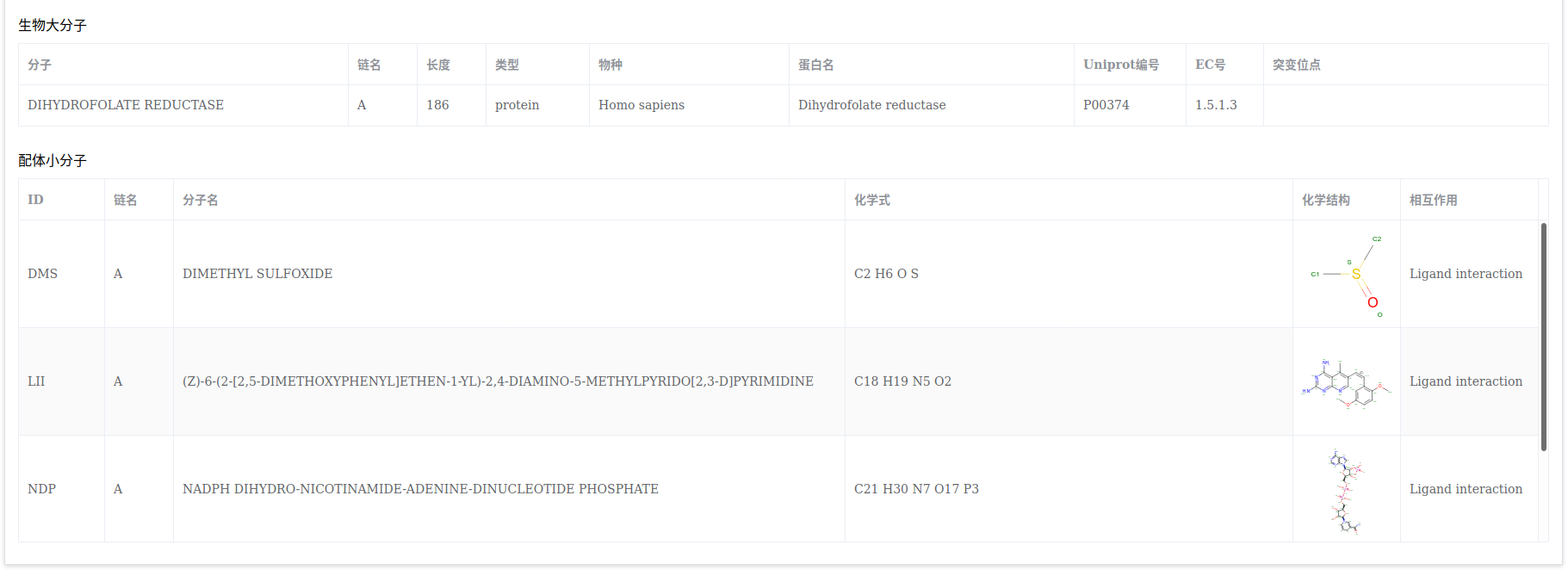
从PDB记录表中查找合适的蛋白结构,必要时,选择该行,点击【查看PDB】了解其结构信息,点击【下载PDB】可下载文件。
本功能主要为分子对接、分子动力学模拟等需要蛋白质三维结构的计算工作而准备。当有多个蛋白PDB结构可供选择时,一般选择含有配体的、分辨率高(数值小)的X射线衍射晶体结构;其次,还可考虑
R(free)和R(work),选择数值较小的。通过缩略图(点击可放大)可大致直观了解蛋白结构情况(比如,是否完整,是否多聚体,是否含有核酸、配体小分子)。举例:假设通过实验发现某个蛋白的抑制剂小分子,拟进行分子对接计算探究其机理。
当可选PDB结构较多时,挑选结构较为完整(可对比
残基数与Uniprot记录中的序列长度)、包含活性位点(查看配体所在位置是否为预期的活性位点)、分辨率最小的晶体结构。若该结构没有配体分子,可另找有配体的晶体结构,了解共晶配体的位置(氨基酸编号)。最后,用分辨率最小的晶体结构做对接,用配体分子或了解到的位置信息定义口袋。注意:某种蛋白可能既有激动剂又有拮抗剂,或者含有多个配体,其中某些只是助溶剂、溶剂分子、金属配合物,要注意分辨共晶配体属于哪一类(查看
标题或点击PDB编号跳转到RCSB PDB网站,查看文献摘要)。